Model Training¶
Onze data is nu klaar om getrained te worden. We kunnen ML toepasssingen opdelen in 2 grote categorien. We zullen gebruik maken van de low code library pycaret om alles te demonstreren.
Unsupervised¶
Unsupervised learning maakt gebruik van machine learning-algoritmen om ongelabelde datasets te analyseren en te clusteren. Deze algoritmen ontdekken verborgen patronen in gegevens zonder dat menselijke tussenkomst nodig is (daarom zijn ze “zonder toezicht”).
Niet-gesuperviseerde leermodellen worden gebruikt voor drie hoofdtaken: clustering, associatie en dimensionaliteitsreductie:
Dimensionaliteitsreductie is een leertechniek die wordt gebruikt wanneer het aantal kenmerken (of dimensies) in een bepaalde dataset te hoog is. Het reduceert het aantal gegevensinvoer tot een beheersbare omvang en behoudt tegelijkertijd de gegevensintegriteit.
Clustering¶
is een dataminingtechniek voor het groeperen van niet-gelabelde gegevens op basis van hun overeenkomsten of verschillen. K-means clusteringalgoritmen wijzen bijvoorbeeld vergelijkbare gegevenspunten toe aan groepen, waarbij de K-waarde de grootte van de groepering en granulariteit vertegenwoordigt. Deze techniek is handig voor marktsegmentatie, beeldcompressie, enz.
from pycaret.datasets import get_data
jewellery = get_data('jewellery')
from pycaret.clustering import *
exp_name = setup(data = jewellery,silent=True)
| Description | Value | |
|---|---|---|
| 0 | session_id | 5828 |
| 1 | Original Data | (505, 4) |
| 2 | Missing Values | False |
| 3 | Numeric Features | 4 |
| 4 | Categorical Features | 0 |
| 5 | Ordinal Features | False |
| 6 | High Cardinality Features | False |
| 7 | High Cardinality Method | None |
| 8 | Transformed Data | (505, 4) |
| 9 | CPU Jobs | -1 |
| 10 | Use GPU | False |
| 11 | Log Experiment | False |
| 12 | Experiment Name | cluster-default-name |
| 13 | USI | 22a9 |
| 14 | Imputation Type | simple |
| 15 | Iterative Imputation Iteration | None |
| 16 | Numeric Imputer | mean |
| 17 | Iterative Imputation Numeric Model | None |
| 18 | Categorical Imputer | mode |
| 19 | Iterative Imputation Categorical Model | None |
| 20 | Unknown Categoricals Handling | least_frequent |
| 21 | Normalize | False |
| 22 | Normalize Method | None |
| 23 | Transformation | False |
| 24 | Transformation Method | None |
| 25 | PCA | False |
| 26 | PCA Method | None |
| 27 | PCA Components | None |
| 28 | Ignore Low Variance | False |
| 29 | Combine Rare Levels | False |
| 30 | Rare Level Threshold | None |
| 31 | Numeric Binning | False |
| 32 | Remove Outliers | False |
| 33 | Outliers Threshold | None |
| 34 | Remove Multicollinearity | False |
| 35 | Multicollinearity Threshold | None |
| 36 | Remove Perfect Collinearity | False |
| 37 | Clustering | False |
| 38 | Clustering Iteration | None |
| 39 | Polynomial Features | False |
| 40 | Polynomial Degree | None |
| 41 | Trignometry Features | False |
| 42 | Polynomial Threshold | None |
| 43 | Group Features | False |
| 44 | Feature Selection | False |
| 45 | Feature Selection Method | classic |
| 46 | Features Selection Threshold | None |
| 47 | Feature Interaction | False |
| 48 | Feature Ratio | False |
| 49 | Interaction Threshold | None |
kmeans = create_model('kmeans')
| Silhouette | Calinski-Harabasz | Davies-Bouldin | Homogeneity | Rand Index | Completeness | |
|---|---|---|---|---|---|---|
| 0 | 0.7207 | 5011.8113 | 0.4114 | 0 | 0 | 0 |
kmeans_df = assign_model(kmeans)
kmeans_df.head()
| Age | Income | SpendingScore | Savings | Cluster | |
|---|---|---|---|---|---|
| 0 | 58 | 77769 | 0.791329 | 6559.829923 | Cluster 2 |
| 1 | 59 | 81799 | 0.791082 | 5417.661426 | Cluster 2 |
| 2 | 62 | 74751 | 0.702657 | 9258.992965 | Cluster 2 |
| 3 | 59 | 74373 | 0.765680 | 7346.334504 | Cluster 2 |
| 4 | 87 | 17760 | 0.348778 | 16869.507130 | Cluster 0 |
plot_model(kmeans,plot= 'distribution')
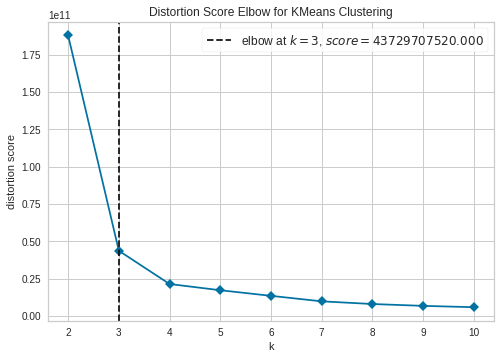
Met de elbow techniek kunnen we het optimaal aantal clusters gaan berekenen.
plot_model(kmeans,plot= 'elbow')
Associatie regels¶
is unsupervised ML techniek die gebruikt wordt om relaties tussen variabelen in een bepaalde dataset te vinden. Deze methoden worden vaak gebruikt voor marktmandanalyse en aanbevelingsengines, in de trant van “Klanten die dit artikel kochten, kochten ook”.
from pycaret.datasets import get_data
data = get_data('france')
from pycaret.arules import *
exp = setup(data = data, transaction_id = 'InvoiceNo', item_id = 'Description')
| InvoiceNo | StockCode | Description | Quantity | InvoiceDate | UnitPrice | CustomerID | Country | |
|---|---|---|---|---|---|---|---|---|
| 0 | 536370 | 22728 | ALARM CLOCK BAKELIKE PINK | 24 | 12/1/2010 8:45 | 3.75 | 12583.0 | France |
| 1 | 536370 | 22727 | ALARM CLOCK BAKELIKE RED | 24 | 12/1/2010 8:45 | 3.75 | 12583.0 | France |
| 2 | 536370 | 22726 | ALARM CLOCK BAKELIKE GREEN | 12 | 12/1/2010 8:45 | 3.75 | 12583.0 | France |
| 3 | 536370 | 21724 | PANDA AND BUNNIES STICKER SHEET | 12 | 12/1/2010 8:45 | 0.85 | 12583.0 | France |
| 4 | 536370 | 21883 | STARS GIFT TAPE | 24 | 12/1/2010 8:45 | 0.65 | 12583.0 | France |
| Description | Value |
|---|---|
| session_id | 7696 |
| # Transactions | 461 |
| # Items | 1565 |
| Ignore Items | None |
rule1 = create_model(metric='confidence', threshold=0.7, min_support=0.05)
plot_model(rule1, plot='3d')
Dimensionaliteitsreductie¶
is een leertechniek die wordt gebruikt wanneer het aantal kenmerken (of dimensies) in een bepaalde dataset te hoog is. Het reduceert het aantal tot een beheersbare omvang en behoudt tegelijkertijd de gegevensintegriteit.
Dimensionaliteitsreductie technieken worden veel gebruikt bij feature engineering.
Curse of dimensionality¶
wanneer de dimensionaliteit toeneemt, neemt het volume van de ruimte zo snel toe dat de beschikbare gegevens schaars worden. Deze schaarste is problematisch voor elke methode die statistische significantie vereist
Supervised¶
Supervised learning is een machine learning-benadering die wordt gedefinieerd door het gebruik van gelabelde datasets. Deze datasets zijn ontworpen om algoritmen te trainen of te “superviseren” om gegevens te classificeren of resultaten nauwkeurig te voorspellen. Met behulp van gelabelde inputs en outputs kan het model zijn nauwkeurigheid meten en in de loop van de tijd leren.
Gesuperviseerd leren kan worden onderverdeeld in twee soorten problemen bij datamining: classificatie en regressie:
Classification¶
is een algoritme om testgegevens nauwkeurig toe te wijzen aan specifieke categorieën, zoals het scheiden van appels van peren.
from pycaret.datasets import get_data
juice = get_data('juice')
from pycaret.classification import *
exp_name = setup(data = juice, silent = True, target = 'Purchase')
| Description | Value | |
|---|---|---|
| 0 | session_id | 6818 |
| 1 | Target | Purchase |
| 2 | Target Type | Binary |
| 3 | Label Encoded | CH: 0, MM: 1 |
| 4 | Original Data | (1070, 19) |
| 5 | Missing Values | False |
| 6 | Numeric Features | 13 |
| 7 | Categorical Features | 5 |
| 8 | Ordinal Features | False |
| 9 | High Cardinality Features | False |
| 10 | High Cardinality Method | None |
| 11 | Transformed Train Set | (748, 17) |
| 12 | Transformed Test Set | (322, 17) |
| 13 | Shuffle Train-Test | True |
| 14 | Stratify Train-Test | False |
| 15 | Fold Generator | StratifiedKFold |
| 16 | Fold Number | 10 |
| 17 | CPU Jobs | -1 |
| 18 | Use GPU | False |
| 19 | Log Experiment | False |
| 20 | Experiment Name | clf-default-name |
| 21 | USI | e349 |
| 22 | Imputation Type | simple |
| 23 | Iterative Imputation Iteration | None |
| 24 | Numeric Imputer | mean |
| 25 | Iterative Imputation Numeric Model | None |
| 26 | Categorical Imputer | constant |
| 27 | Iterative Imputation Categorical Model | None |
| 28 | Unknown Categoricals Handling | least_frequent |
| 29 | Normalize | False |
| 30 | Normalize Method | None |
| 31 | Transformation | False |
| 32 | Transformation Method | None |
| 33 | PCA | False |
| 34 | PCA Method | None |
| 35 | PCA Components | None |
| 36 | Ignore Low Variance | False |
| 37 | Combine Rare Levels | False |
| 38 | Rare Level Threshold | None |
| 39 | Numeric Binning | False |
| 40 | Remove Outliers | False |
| 41 | Outliers Threshold | None |
| 42 | Remove Multicollinearity | False |
| 43 | Multicollinearity Threshold | None |
| 44 | Remove Perfect Collinearity | True |
| 45 | Clustering | False |
| 46 | Clustering Iteration | None |
| 47 | Polynomial Features | False |
| 48 | Polynomial Degree | None |
| 49 | Trignometry Features | False |
| 50 | Polynomial Threshold | None |
| 51 | Group Features | False |
| 52 | Feature Selection | False |
| 53 | Feature Selection Method | classic |
| 54 | Features Selection Threshold | None |
| 55 | Feature Interaction | False |
| 56 | Feature Ratio | False |
| 57 | Interaction Threshold | None |
| 58 | Fix Imbalance | False |
| 59 | Fix Imbalance Method | SMOTE |
best_model = compare_models()
| Model | Accuracy | AUC | Recall | Prec. | F1 | Kappa | MCC | TT (Sec) | |
|---|---|---|---|---|---|---|---|---|---|
| lda | Linear Discriminant Analysis | 0.8423 | 0.9068 | 0.8062 | 0.8011 | 0.8001 | 0.6700 | 0.6742 | 0.0150 |
| lr | Logistic Regression | 0.8409 | 0.9077 | 0.7723 | 0.8193 | 0.7913 | 0.6632 | 0.6679 | 0.2280 |
| ridge | Ridge Classifier | 0.8409 | 0.0000 | 0.8028 | 0.8004 | 0.7978 | 0.6669 | 0.6713 | 0.0080 |
| gbc | Gradient Boosting Classifier | 0.8208 | 0.8948 | 0.7689 | 0.7788 | 0.7712 | 0.6242 | 0.6269 | 0.0820 |
| ada | Ada Boost Classifier | 0.8088 | 0.8840 | 0.7280 | 0.7773 | 0.7463 | 0.5939 | 0.6002 | 0.0650 |
| rf | Random Forest Classifier | 0.8007 | 0.8828 | 0.7485 | 0.7495 | 0.7466 | 0.5826 | 0.5851 | 0.4550 |
| lightgbm | Light Gradient Boosting Machine | 0.7994 | 0.8761 | 0.7385 | 0.7559 | 0.7439 | 0.5795 | 0.5829 | 0.0400 |
| et | Extra Trees Classifier | 0.7834 | 0.8465 | 0.7084 | 0.7363 | 0.7205 | 0.5440 | 0.5458 | 0.4540 |
| dt | Decision Tree Classifier | 0.7687 | 0.7629 | 0.7147 | 0.7074 | 0.7076 | 0.5167 | 0.5204 | 0.0150 |
| nb | Naive Bayes | 0.7633 | 0.8400 | 0.7892 | 0.6811 | 0.7252 | 0.5207 | 0.5322 | 0.0080 |
| knn | K Neighbors Classifier | 0.7045 | 0.7532 | 0.6057 | 0.6344 | 0.6150 | 0.3762 | 0.3800 | 0.1200 |
| svm | SVM - Linear Kernel | 0.5643 | 0.0000 | 0.2033 | 0.1787 | 0.1194 | 0.0040 | 0.0142 | 0.0120 |
| qda | Quadratic Discriminant Analysis | 0.4663 | 0.5383 | 0.8752 | 0.4325 | 0.5674 | 0.0737 | 0.0756 | 0.0100 |
Metrics
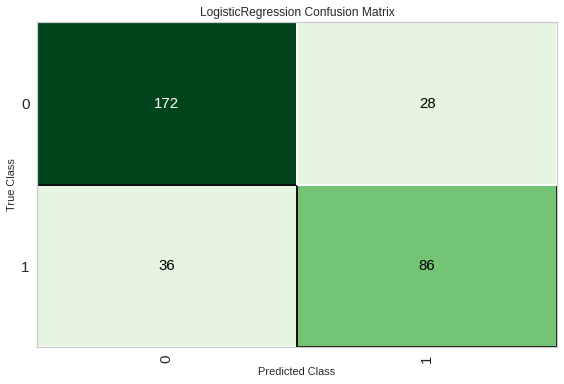
Als we de voorspelde resulataten van een model hebben dan kunnen we deze vergelijken met de effectieve waarden. Deze metrics geven ons inzichten over het model. We proberen altijd ten opzichten van een bepaalde metric te gaan optimaliseren. Omdat alle metrics wel een iets ander aspect van het model accentueren. Bij classificatie kijken we vooral naar de confusion matrix.
lr = create_model('lr')
plot_model(lr,'confusion_matrix')
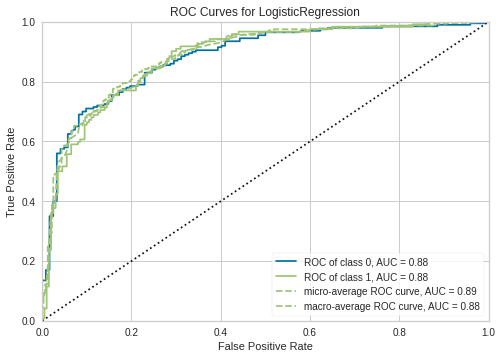
Performance metrics zijn dan een specifieke combinatie van deze TP FP TN FN.
Precision = TP/(TP+FP)
plot_model(lr)
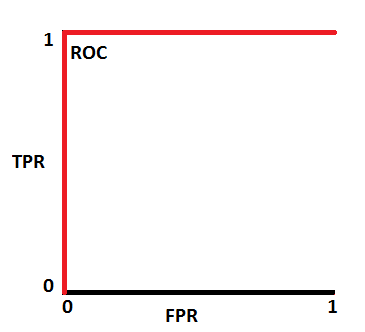
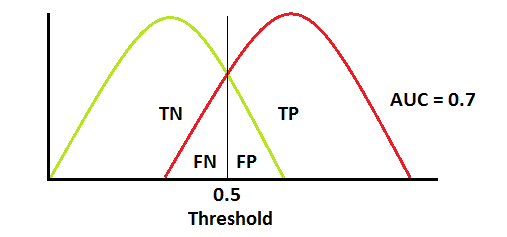
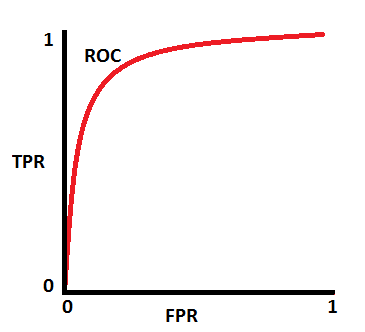
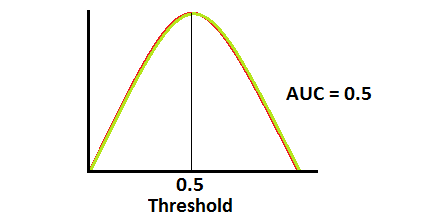
Regressie¶
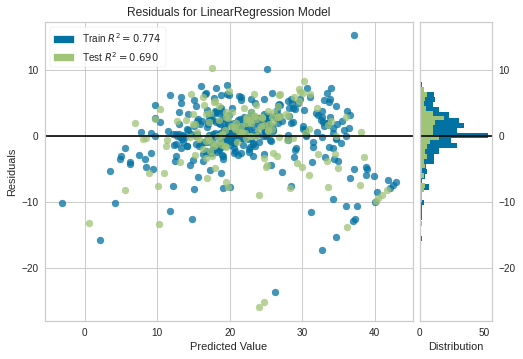
Regressiemodellen zijn handig voor het voorspellen van numerieke waarden op basis van verschillende gegevenspunten, zoals projecties van verkoopopbrengsten voor een bepaald bedrijf.Regressie is een ander type begeleide leermethode die een algoritme gebruikt om de relatie tussen afhankelijke en onafhankelijke variabelen te begrijpen. Regressiemodellen zijn handig voor het voorspellen van numerieke waarden op basis van verschillende gegevenspunten, zoals projecties van verkoopopbrengsten voor een bepaald bedrijf. Enkele populaire regressie-algoritmen zijn lineaire regressie, logistische regressie en polynomiale regressie.
from pycaret.datasets import get_data
boston = get_data('boston')
from pycaret.regression import *
exp_name = setup(data = boston, silent=True , target = 'medv')
| Description | Value | |
|---|---|---|
| 0 | session_id | 4671 |
| 1 | Target | medv |
| 2 | Original Data | (506, 14) |
| 3 | Missing Values | False |
| 4 | Numeric Features | 11 |
| 5 | Categorical Features | 2 |
| 6 | Ordinal Features | False |
| 7 | High Cardinality Features | False |
| 8 | High Cardinality Method | None |
| 9 | Transformed Train Set | (354, 21) |
| 10 | Transformed Test Set | (152, 21) |
| 11 | Shuffle Train-Test | True |
| 12 | Stratify Train-Test | False |
| 13 | Fold Generator | KFold |
| 14 | Fold Number | 10 |
| 15 | CPU Jobs | -1 |
| 16 | Use GPU | False |
| 17 | Log Experiment | False |
| 18 | Experiment Name | reg-default-name |
| 19 | USI | 47fd |
| 20 | Imputation Type | simple |
| 21 | Iterative Imputation Iteration | None |
| 22 | Numeric Imputer | mean |
| 23 | Iterative Imputation Numeric Model | None |
| 24 | Categorical Imputer | constant |
| 25 | Iterative Imputation Categorical Model | None |
| 26 | Unknown Categoricals Handling | least_frequent |
| 27 | Normalize | False |
| 28 | Normalize Method | None |
| 29 | Transformation | False |
| 30 | Transformation Method | None |
| 31 | PCA | False |
| 32 | PCA Method | None |
| 33 | PCA Components | None |
| 34 | Ignore Low Variance | False |
| 35 | Combine Rare Levels | False |
| 36 | Rare Level Threshold | None |
| 37 | Numeric Binning | False |
| 38 | Remove Outliers | False |
| 39 | Outliers Threshold | None |
| 40 | Remove Multicollinearity | False |
| 41 | Multicollinearity Threshold | None |
| 42 | Remove Perfect Collinearity | True |
| 43 | Clustering | False |
| 44 | Clustering Iteration | None |
| 45 | Polynomial Features | False |
| 46 | Polynomial Degree | None |
| 47 | Trignometry Features | False |
| 48 | Polynomial Threshold | None |
| 49 | Group Features | False |
| 50 | Feature Selection | False |
| 51 | Feature Selection Method | classic |
| 52 | Features Selection Threshold | None |
| 53 | Feature Interaction | False |
| 54 | Feature Ratio | False |
| 55 | Interaction Threshold | None |
| 56 | Transform Target | False |
| 57 | Transform Target Method | box-cox |
best_model = compare_models()
| Model | MAE | MSE | RMSE | R2 | RMSLE | MAPE | TT (Sec) | |
|---|---|---|---|---|---|---|---|---|
| et | Extra Trees Regressor | 2.0090 | 8.8154 | 2.8263 | 0.8924 | 0.1368 | 0.1053 | 0.4060 |
| gbr | Gradient Boosting Regressor | 2.1695 | 11.0702 | 3.1526 | 0.8626 | 0.1466 | 0.1128 | 0.0690 |
| lightgbm | Light Gradient Boosting Machine | 2.2775 | 12.0425 | 3.2793 | 0.8566 | 0.1475 | 0.1142 | 0.0250 |
| rf | Random Forest Regressor | 2.2614 | 13.1724 | 3.4452 | 0.8392 | 0.1548 | 0.1175 | 0.4520 |
| ada | AdaBoost Regressor | 2.7565 | 16.8872 | 3.8880 | 0.7911 | 0.1816 | 0.1465 | 0.0650 |
| lr | Linear Regression | 3.2293 | 21.7648 | 4.5754 | 0.7281 | 0.2445 | 0.1669 | 0.0140 |
| lar | Least Angle Regression | 3.2293 | 21.7647 | 4.5754 | 0.7281 | 0.2445 | 0.1669 | 0.0160 |
| ridge | Ridge Regression | 3.2081 | 21.8628 | 4.5771 | 0.7276 | 0.2360 | 0.1663 | 0.0080 |
| br | Bayesian Ridge | 3.2143 | 22.2970 | 4.6190 | 0.7227 | 0.2347 | 0.1672 | 0.0090 |
| huber | Huber Regressor | 3.2550 | 25.7317 | 4.9230 | 0.6871 | 0.2537 | 0.1676 | 0.0250 |
| dt | Decision Tree Regressor | 3.0648 | 24.9198 | 4.7315 | 0.6855 | 0.2092 | 0.1584 | 0.0110 |
| en | Elastic Net | 3.5718 | 26.6459 | 5.0624 | 0.6668 | 0.2520 | 0.1770 | 0.0090 |
| lasso | Lasso Regression | 3.6084 | 26.9616 | 5.0859 | 0.6639 | 0.2529 | 0.1782 | 0.0080 |
| omp | Orthogonal Matching Pursuit | 3.8332 | 28.6061 | 5.2526 | 0.6460 | 0.3033 | 0.2015 | 0.0090 |
| knn | K Neighbors Regressor | 4.3839 | 41.0976 | 6.3080 | 0.4738 | 0.2472 | 0.2068 | 0.0650 |
| llar | Lasso Least Angle Regression | 6.5724 | 82.2513 | 9.0062 | -0.0309 | 0.3879 | 0.3601 | 0.0080 |
| par | Passive Aggressive Regressor | 7.0552 | 90.2726 | 9.3364 | -0.1480 | 0.4519 | 0.3611 | 0.0170 |
lr = create_model('lr')
| MAE | MSE | RMSE | R2 | RMSLE | MAPE | |
|---|---|---|---|---|---|---|
| 0 | 3.1475 | 20.8296 | 4.5639 | 0.7625 | 0.1866 | 0.1482 |
| 1 | 3.3793 | 25.8070 | 5.0801 | 0.6748 | 0.2576 | 0.1902 |
| 2 | 3.2143 | 19.9601 | 4.4677 | 0.7849 | 0.2241 | 0.1753 |
| 3 | 2.7186 | 12.9753 | 3.6021 | 0.7453 | 0.1720 | 0.1369 |
| 4 | 3.5540 | 25.8130 | 5.0807 | 0.6424 | 0.2906 | 0.1703 |
| 5 | 4.4675 | 44.4164 | 6.6646 | 0.5995 | 0.5755 | 0.2171 |
| 6 | 2.8228 | 15.1059 | 3.8866 | 0.8536 | 0.1784 | 0.1404 |
| 7 | 3.0503 | 21.2718 | 4.6121 | 0.7677 | 0.1740 | 0.1319 |
| 8 | 2.4780 | 10.0045 | 3.1630 | 0.8165 | 0.1688 | 0.1362 |
| 9 | 3.4606 | 21.4647 | 4.6330 | 0.6344 | 0.2173 | 0.2224 |
| Mean | 3.2293 | 21.7648 | 4.5754 | 0.7281 | 0.2445 | 0.1669 |
| SD | 0.5238 | 9.0011 | 0.9115 | 0.0808 | 0.1169 | 0.0322 |
plot_model(lr)
Models¶
We gaan even kort schetsen hoe de volgende modellen werken.
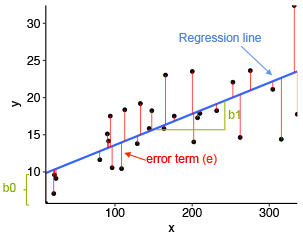
Lineaire modellen¶
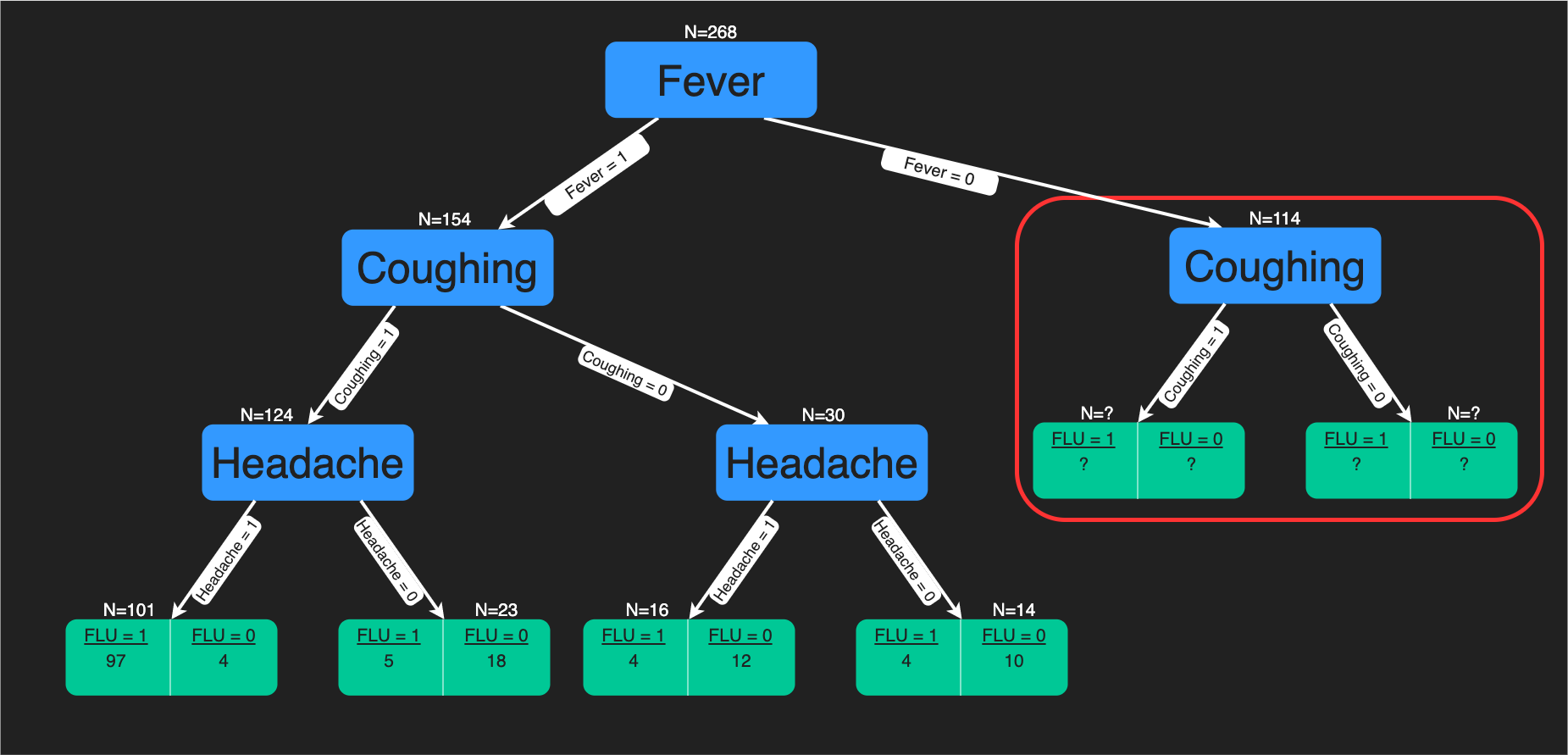
Decision trees¶
Hyperparameters¶
Hyperparameters zij de parameters die we meegeven bij de start van een model training. Bij een decisiontree is dat het aantal splits die we maximaal gaan maken.
Ensembles¶
We kunnen ook verschillende modellen met elkaar gaan combineren en zo naar een nog optimalere oplossing gaan zoeken. We nemen verschillende zwakke modellen en gaan die dan combineren om tot betere performantie te komen.
Stacking¶
Dit is verschillende modellen (SVM,Random forrest) met elkaar gaan combineren en een metamodel bovenop de andere modellen gaan trainen om te gaan beslissen welk model we gaan gebruiken.
Bagging¶
Werkt met parallele weak learners en deze dan combineren door een gemiddelde te nemen over alle zwakke modellen.
RandomForrest
Boosting¶
Bij deze techniek gaan we zwakke modellen sequentieel achter elkaar gaan zetten.
Catboost
XGBoost
from pycaret.datasets import get_data
diabetes = get_data('diabetes')
# Importing module and initializing setup
from pycaret.classification import *
clf1 = setup(data = diabetes,silent= True, target = 'Class variable')
# train a decision tree model
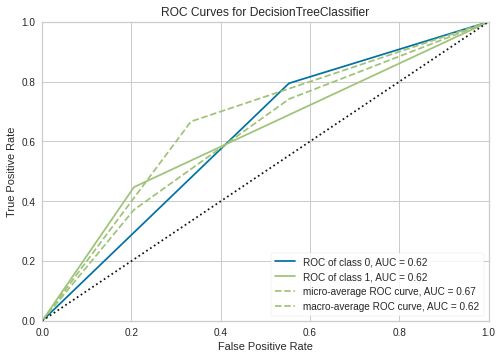
dt = create_model('dt')
# train a bagging classifier on dt
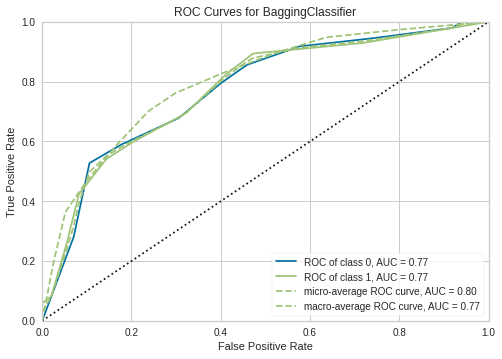
bagged_dt = ensemble_model(dt, method = 'Bagging')
# train a adaboost classifier on dt with 100 estimators
boosted_dt = ensemble_model(dt, method = 'Boosting', n_estimators = 100)
| Accuracy | AUC | Recall | Prec. | F1 | Kappa | MCC | |
|---|---|---|---|---|---|---|---|
| 0 | 0.6296 | 0.5694 | 0.3889 | 0.4375 | 0.4118 | 0.1429 | 0.1434 |
| 1 | 0.7593 | 0.7222 | 0.6111 | 0.6471 | 0.6286 | 0.4507 | 0.4511 |
| 2 | 0.7593 | 0.6944 | 0.5000 | 0.6923 | 0.5806 | 0.4179 | 0.4288 |
| 3 | 0.6852 | 0.6528 | 0.5556 | 0.5263 | 0.5405 | 0.3014 | 0.3016 |
| 4 | 0.7407 | 0.6917 | 0.5263 | 0.6667 | 0.5882 | 0.4028 | 0.4088 |
| 5 | 0.6667 | 0.6466 | 0.5789 | 0.5238 | 0.5500 | 0.2863 | 0.2872 |
| 6 | 0.6667 | 0.5865 | 0.3158 | 0.5455 | 0.4000 | 0.1913 | 0.2050 |
| 7 | 0.6792 | 0.6357 | 0.5000 | 0.5294 | 0.5143 | 0.2751 | 0.2754 |
| 8 | 0.6981 | 0.6905 | 0.6667 | 0.5455 | 0.6000 | 0.3614 | 0.3661 |
| 9 | 0.6981 | 0.6770 | 0.6111 | 0.5500 | 0.5789 | 0.3447 | 0.3458 |
| Mean | 0.6983 | 0.6567 | 0.5254 | 0.5664 | 0.5393 | 0.3175 | 0.3213 |
| SD | 0.0406 | 0.0465 | 0.1011 | 0.0741 | 0.0733 | 0.0934 | 0.0935 |
plot_model(dt)
plot_model(bagged_dt)
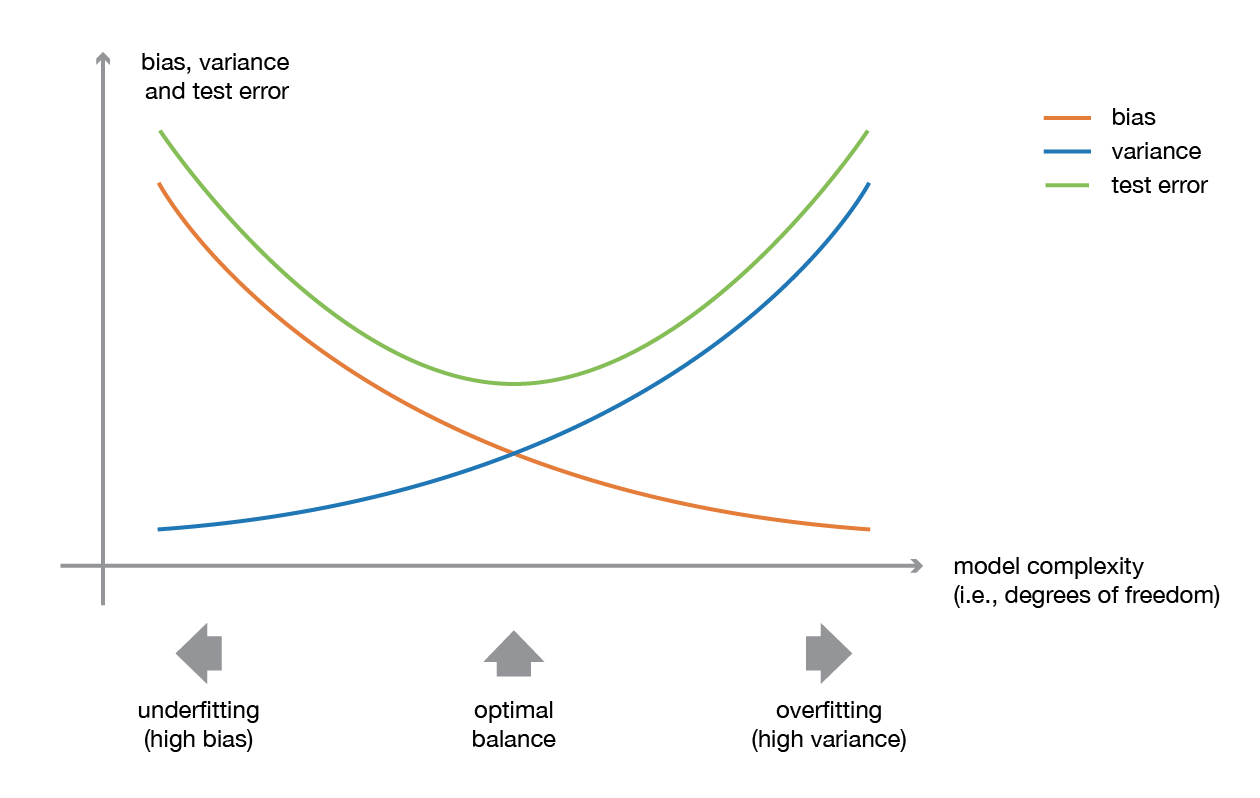
Overfitting¶
We hebben het fenomeen overfitting al bekeken en technieken gevonden om overfitting te vermijden. Er zijn nog andere manieren zoals regularisatie bij lineaire modellen.
Lasso
Ridge
Elasticnet
